基因组组区重叠Venn图
简介
默认最多绘制6个结果的交集venn图
数据说明
数据默认包括3列:染色体,开始,结束
若要考虑正负链,则包括6列:chrom start end name value strand
若输入数据集为A和B,那么A特异的就是10_A.bed,B特异的就是01_B.bed,重叠的为11_A_B.bed,其中A特异的和重叠的结果均以A中的区域表示。由于可能存在多对多的现象,因此图中的数字加起来可能与输入的行数不一致。调用intervenn。
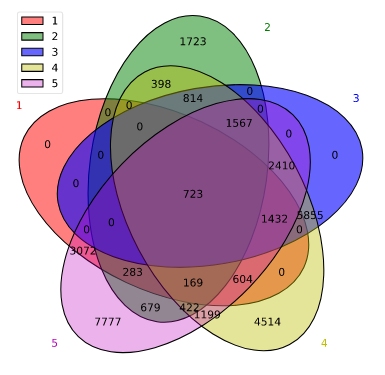
论文例子
Intervene: a tool for intersection and visualization of multiple gene or genomic region sets Fig 2A.
| 输入 |
chr1 30 50
chr2 30 50 |
| 输出 |
|
如何引用?
建议直接写网址。助力发表9000+篇
(google学术),7400+篇
(知网)论文
正式引用:Tang D, Chen M, Huang X, Zhang G, Zeng L, Zhang G, Wu S, Wang Y.
SRplot: A free online platform for data visualization and graphing. PLoS One. 2023 Nov 9;18(11):e0294236. doi: 10.1371/journal.pone.0294236. PMID: 37943830.
方法章节:Heatmap was plotted by https://www.bioinformatics.com.cn (last accessed on 10 Dec 2024), an online platform for data analysis and visualization.
致谢章节:We thank Mingjie Chen (Shanghai NewCore Biotechnology Co., Ltd.) for providing data analysis and visualization support.


